Nội dung toàn văn Tiêu chuẩn quốc gia TCVN 10781:2015 (ISO/TS 13136:2012) về Vi sinh vật trong thực phẩm và thức ăn chăn nuôi – Phương pháp phát hiện vi sinh vật gây bệnh trong thực phẩm bằng phản ứng chuỗi polymerase (PCR) thời gian thực – Phát hiện Escherichia coli sinh độc tố Shiga (STEC) và xác định các nhóm huyết thanh O157, O111, O26,O103 và O145
TIÊU CHUẨN QUỐC GIA
TCVN 10781:2015
ISO/TS 13136:2012
VI SINH VẬT TRONG THỰC PHẨM VÀ THỨC ĂN CHĂN NUÔI – PHƯƠNG PHÁP PHÁT HIỆN VI SINH VẬT GÂY BỆNH TRONG THỰC PHẨM BẰNG PHẢN ỨNG CHUỖI POLYMERASE (PCR) THỜI GIAN THỰC – PHÁT HIỆN ESCHERICHIA COLI SINH ĐỘC TỐ SHIGA (STEC) VÀ XÁC ĐỊNH CÁC NHÓM HUYẾT THANH O157, O111, O26, O103 VÀ O145
Microbiology of food and animal feed – Real-time polymerase chain reaction (PCR)-based method for the detection of food-borne pathogens – Horizontal method for the detection of Shiga toxin-producing Escherichia coli (STEC) and the determination of O157, O111, O26, O103 and O145 serogroups
Lời nói đầu
TCVN 10781:2015 hoàn toàn tương đương với ISO/TS 13136:2012;
TCVN 10781:2015 do Ban kỹ thuật tiêu chuẩn quốc gia TCVN/TC/F13 Phương pháp phân tích và lấy mẫu biên soạn, Tổng cục Tiêu chuẩn Đo lường Chất lượng thẩm định, Bộ Khoa học và Công nghệ công bố.
Lời giới thiệu
Escherichia coli sinh độc tố Shiga (STEC) là E. coli gây bệnh có thể gây tiêu chảy cũng như các bệnh nghiêm trọng hơn ở người như xuất huyết đại tràng và hội chứng ure huyết cao-tán huyết (HUS). Mặc dù STEC có thể thuộc về một lượng lớn các nhóm huyết thanh, các nhóm huyết thanh này liên kết vững chắc với phần lớn các dạng bệnh, cụ thể là HUS, thuộc nhóm huyết thanh O157, O111, O103 và O145 (Tài liệu tham khảo [1]).
Tiêu chuẩn này sử dụng các danh pháp dưới đây:
– stx: các gen sinh độc tố Shiga (đồng nghĩa với vxt);
– Stx: độc tố Shiga (đồng nghĩa với Vtx: Verocytotoxin);
– STEC: Escherichia coli sinh độc tố Shiga (đồng nghĩa với VTEC: Escherichia coli sinh Verocytotoxin).
VI SINH VẬT TRONG THỰC PHẨM VÀ THỨC ĂN CHĂN NUÔI – PHƯƠNG PHÁP PHÁT HIỆN VI SINH VẬT GÂY BỆNH TRONG THỰC PHẨM BẰNG PHẢN ỨNG CHUỖI POLYMERASE (PCR) THỜI GIAN THỰC – PHÁT HIỆN ESCHERICHIA COLI SINH ĐỘC TỐ SHIGA (STEC) VÀ XÁC ĐỊNH CÁC NHÓM HUYẾT THANH O157, O111, O26, O103 VÀ O145
Microbiology of food and animal feed – Real-time polymerase chain reaction (PCR)-based method for the detection of food-borne pathogens – Horizontal method for the detection of Shiga toxin-producing Escherichia coli (STEC) and the determination of O157, O111, O26, O103 and O145 serogroups
CHÚ Ý – Cần xem xét mọi STEC có khả năng gây bệnh cho người và có khả năng gây bệnh nghiêm trọng phụ thuộc vào hồ sơ nguy cơ của thực phẩm (thực phẩm ăn liền với thực phẩm được tiêu thụ sau khi xử lý công nghệ như tiệt trùng, nấu chín v.v… để giảm thiểu vi khuẩn có trong thực phẩm) và tình trạng sức khỏe của người tiêu thụ thực phẩm đó.
Ngoài ra, các loài vi khuẩn này có bộ gen với tính mềm dẻo cao, có thể có bố trí khác lạ của gen độc có khả năng làm tăng các nhóm huyết thanh gây bệnh mới như E. coli O104 sinh độc tố Shiga gây bệnh dính ruột làm xảy ra dịch HUS bùng phát ở Đức và Pháp vào tháng 5 và tháng 6 năm 2011. E. coli thuộc các nhóm có kiểu huyết thanh không điển hình mới có thể sinh ra từ một thực khuẩn stx-cải biến bởi một chủng E. coli thuộc các nhóm gây bệnh khác với STEC.
Các chủng không điển hình đó nằm trong phạm vi áp dụng của tiêu chuẩn này và có thể phát hiện được vì chúng khẳng định sự có mặt của các gen stx.
1. Phạm vi áp dụng
Tiêu chuẩn này quy định việc nhận biết Escherichia coli sinh độc tố Shiga (STEC) bằng cách phát hiện các gen dưới đây:
a) các gen độc chính của STEC, stx và eae (Tài liệu tham khảo [2][3]);
b) các gen liên quan đến các nhóm huyết thanh O157, O111, O26, O103 và O145 (Tài liệu tham khảo [3][4]).
Trong mọi trường hợp, khi một hoặc cả hai gen stx được phát hiện thì cần phân lập chủng.
Việc phân lập STEC từ các mẫu khẳng định dương tính với sự có mặt của các gen quy định các nhóm huyết thanh trong phạm vi áp dụng của tiêu chuẩn này có thể được thực hiện dễ dàng bằng cách sử dụng các kỹ thuật tăng sinh nhóm huyết thanh đặc hiệu [ví dụ: kỹ thuật phân tách miễn dịch-từ tính (immunomagnetic separation-IMS)].
Nguyên tắc sử dụng phản ứng chuỗi polymerase thời gian thực (real-time PCR) là công nghệ chuẩn để phát hiện các gen độc và các gen liên kết nhóm huyết thanh.
Tiêu chuẩn này áp dụng cho:
1) các sản phẩm dùng cho người và dùng làm thức ăn chăn nuôi;
2) các mẫu môi trường trong khu vực sản xuất và chế biến thực phẩm;
3) các mẫu môi trường trong khu vực sản xuất ban đầu.
2. Tài liệu viện dẫn
Các tài liệu viện dẫn sau rất cần thiết cho việc áp dụng tiêu chuẩn này. Đối với các tài liệu viện dẫn ghi năm công bố thì áp dụng phiên bản được nêu. Đối với các tài liệu viện dẫn không ghi năm công bố thì áp dụng phiên bản mới nhất, bao gồm cả các sửa đổi, bổ sung (nếu có).
TCVN 6404 (ISO 7218), Vi sinh vật trong thực phẩm và thức ăn chăn nuôi – Yêu cầu chung và hướng dẫn kiểm tra vi sinh vật.
TCVN 7682 (ISO 20838), Vi sinh vật trong thực phẩm và thức ăn chăn nuôi – Phản ứng chuỗi polymeraza (PCR) để phát hiện sinh vật gây bệnh từ thực phẩm – Yêu cầu về khuếch đại và phát hiện đối với các phương pháp định tính.
ISO 22174, Microbiology of food and animal feeding stuffs – Polymerase chain reaction (PCR) for the detection of food-borne pathogens – General requirements and definitions (Vi sinh vật trong thực phẩm và thức ăn chăn nuôi – Phản ứng chuỗi trùng hợp để phát hiện sinh vật gây bệnh từ thực phẩm – Yêu cầu chung và định nghĩa).
3. Thuật ngữ và định nghĩa
Trong tiêu chuẩn này sử dụng các thuật ngữ và định nghĩa sau:
3.1. Escherichia coli sinh độc tố Shiga (Shiga toxin-producing Escherichia coli)
STEC
Các chủng E. coli có chứa các gen mã hóa Stx.
3.2. Escherichia coli sinh độc tố Shiga gây tổn thương kết dính và phá hủy/STEC gây tổn thương dạng kết dính và phá hủy (Shiga toxin-producing Escherichia coli causing the attaching and effacing lesion)/(STEC causing attaching and effacing lesion)
Các chủng E. coli có các gen mã hóa Stx và các gen eae mã hóa intimin.
CHÚ THÍCH: Sự kết hợp này của các gen độc là thường liên quan đến hầu hết với các dạng bệnh nghiêm trọng gây ra bởi STEC.
3.3 .Escherichia coli sinh độc tố Shiga thuộc các nhóm huyết thanh có khả năng gây bệnh cao/STEC thuộc các nhóm huyết thanh có khả năng gây bệnh cao (Shiga toxin-producing Escherichia coli belonging to highly pathogenic serogroups)/(STEC belonging to highly pathogenic serogroups)
Các chủng E. coli có các gen mã hóa Stx, gen eae mã hóa intimin và thuộc một trong các nhóm huyết thanh O157, O111, O26, O103 và O145.
CHÚ THÍCH Các định nghĩa từ 3.1 đến 3.3 được tổng hợp từ các dữ liệu dịch tễ học về bệnh gây ra bởi STEC do các tổ chức như Trung tâm Kiểm soát Dịch bệnh Hoa Kỳ quản lý, tại Châu Âu do Trung tâm Phòng chống và Kiểm soát Dịch bệnh Châu Âu và Cơ quan An toàn Thực phẩm Châu Âu quản lý.
4. Nguyên tắc
4.1. Yêu cầu chung
Phương pháp quy định bao gồm các bước trình tự như sau:
a) tăng sinh vi khuẩn;
b) tách chiết axit nucleic;
c) phát hiện các gen độc;
d) phát hiện các gen liên quan đến nhóm huyết thanh;
e) phân lập từ các mẫu dương tính. Hình A.1 là sơ đồ quy trình sàng lọc.
4.2. Tăng sinh vi khuẩn
Tăng số lượng các tế bào STEC cần phát hiện bằng cách ủ phần mẫu thử trong môi trường dinh dưỡng lỏng không chọn lọc được chọn từ:
a) canh thang trypton-đậu tương cải biến (canh thang trypton-đậu tương có bổ sung muối mật Số 3 1,5 g/l, mTSB) có bổ sung novobiocin (mTSB+N) 16 mg/l.
b) nước đệm pepton (BPW);
c) canh thang trypton-đậu tương cải biến (canh thang trypton-đậu tương có bổ sung muối mật Số 3 1,5 g/l, mTSB) có bổ sung acriflavin (mTSB+A) 12 mg/l để phân tích sữa và các sản phẩm sữa.
Cần sử dụng mTSB khi phân tích các nền mẫu nghi ngờ có mức vi sinh vật nhiễm bẩn cao. Novobiocin và acriflavin ức chế sự phát triển của các vi khuẩn Gram dương và thúc đẩy sự phát triển của tế bào Gram âm, bao gồm cả STEC. BPW được sử dụng để phân tích các mẫu được coi là có chứa vi khuẩn đích bị ức chế (ví dụ: các sản phẩm đông lạnh) để phục hồi các tế bào STEC bị ức chế và các mẫu dự kiến mức vi sinh vật nhiễm bẩn thấp hơn mức vi sinh vật nhiễm bẩn trong mẫu tươi.
CHÚ THÍCH: Việc bổ sung novobiocin còn gây tranh cãi và đã được nhiều tác giả nghiên cứu. Thực tế quan sát cho thấy nồng độ ức chế tối thiểu của kháng sinh đối với STEC không phải O157 là thấp hơn so với các chủng O157 (Tài liệu tham khảo [5]). Việc bổ sung novobiocin vào mTSB tăng sinh ở nồng độ thông thường 20 mg/ml, như quy định trong TCVN 7686 (ISO 16654)[19] dường như làm ức chế sự phát triển khoảng một phần ba các chủng không phải O157 (Tài liệu tham khảo [6]) làm tăng nguy cơ cho kết quả âm tính giả.
4.3. Tách chiết axit nucleic
Tách chiết axit nucleic theo các yêu cầu của hệ thống phát hiện đã sử dụng.
4.4. Gen đích
Axit nucleic đã tinh sạch được sử dụng để phát hiện các gen đích dưới đây:
– các gen độc chính của STEC: gen stx, mã hóa các độc tố Shiga và gen eae, mã hóa protein có khối lượng 90 kDa, intimin liên quan đến sự kết dính và cơ chế phá hủy sự kết dính, đặc điểm điển hình của các chủng STEC gây bệnh. Các gen stx mã hóa một họ các độc tố bao gồm hai kiểu chính: stx1 và stx2. stx2 gồm bảy biến thể được công nhận (từ stx2a đến stx2g) (Tài liệu tham khảo [22]). Chỉ có các biến thể stx2a, stx2b, stx2c được tìm thấy là được sinh ra bởi các chủng STEC nêu trong Điều 1, vì vậy tạo thành các gen mã hóa Stx đích nêu trong tiêu chuẩn này. Số lượng bổ sung vào Ngân hàng Gen tương ứng với các gen mã hóa biến thể stx2 là:
– stx2a: X07865
– stx2b: L11078
– stx2c: M59432
– gen eae mã hóa intimin.
– các gen rfbE(O157), wbdl(O111), wzx(O26), ihp1(O145) và wzx(O103) được nhận biết theo các nhóm huyết thanh tương ứng.
4.5. Phát hiện
Tiến hành phát hiện các gen đích theo hệ thống phát hiện đã được sử dụng.
4.6. Phân lập
Nếu nghi ngờ có mặt STEC thì tiến hành phân lập. Nếu phát hiện có một trong các nhóm huyết thanh được quy định trong phạm vi áp dụng của tiêu chuẩn này thì tiến hành tăng sinh nhóm huyết thanh đặc hiệu (ví dụ: IMS) bằng cách nuôi cấy trên thạch trypton-mật-glucuronic (TBX) hoặc môi trường chọn lọc đặc trưng nếu có (xem Phụ lục F, Chú thích 2 và Chú thích 3) để tạo thuận tiện cho việc phân lập STEC ra khỏi hệ vi sinh vật nền.
5. Dịch pha loãng, môi trường nuôi cấy và thuốc thử
Chỉ sử dụng thuốc thử loại tinh khiết phân tích và nước cất vô trùng hoặc nước đã khử khoáng hoặc nước có chất lượng tương đương, trừ khi có quy định khác.
5.1. Môi trường nuôi cấy
5.1.1. Canh thang trypton-đậu tương cải biến (mTSB)
5.1.1.1. Môi trường cơ bản
Thành phần và pH
Sản phẩm phân hủy từ casein bằng enzym 17 g
Sản phẩm phân hủy từ đậu tương bằng enzym 3 g
D(+)-Glucose 2,5 g
Natri clorua 5 g
Dikali hydrophosphat (K2HPO4) 4 g
Muối mật Số 3 1,5 g
Nước đến 1 000 ml
pH 7,4 ± 0,2
Chuẩn bị
Hòa tan các thành phần hoặc môi trường khan trong nước. Dùng máy đo pH, chỉnh pH đến pH 7,4 ± 0,2 ở 25 oC và khử trùng bằng nồi hấp ở 121 oC trong 15 min.
5.1.1.2. Dung dịch novobiocin
Thành phần
Novobiocin 0,16 g
Nước 10 ml
Chuẩn bị
Hòa tan novobiocin trong nước và khử trùng bằng cách lọc qua màng lọc cỡ lỗ 0,22 μm hoặc 0,45 μm.
Chuẩn bị dung dịch này trong ngày sử dụng.
5.1.1.3. Dung dịch acriflavin
Thành phần
Acriflavin 0,12 g
Nước 10 ml
Chuẩn bị
Hòa tan acriflavin trong nước và khử trùng bằng cách lọc qua màng lọc cỡ lỗ 0,22 μm hoặc 0,45 μm.
Chuẩn bị dung dịch này trong ngày sử dụng.
5.1.1.4. Chuẩn bị môi trường hoàn chỉnh
Ngay trước khi sử dụng, cho 1 ml dung dịch novobiocin (5.1.1.2) hoặc dung dịch acriflavin (5.1.1.3) vào 1 000 ml mTSB (5.1.1.1) đã làm lạnh.
Nồng độ cuối của novobiocin phải là 16 mg/l mTSB.
Nồng độ cuối của acriflavin phải là 12 mg/l mTSB.
5.1.2. Nước đệm pepton (BPW) Thành phần và pH
Pepton 10 g
Natri clorua 5,0 g
Dinatri phosphat (Na2HPO4) 3,5 g
Kali dihydrophosphat (KH2PO4) 1,5 g
Nước đến 1 000 ml
pH 7,0 ± 0,2
Chuẩn bị
Hòa tan các thành phần hoặc bột khô trong nước. Dùng máy đo pH, chỉnh pH đến pH 7,0 ± 0,2 ở 25 oC, sử dụng bằng máy đo pH và tiệt trùng bằng nồi hấp ở 121 oC trong 15 min.
5.2. Thuốc thử dùng để tách chiết axit nucleic
Thuốc thử dùng để tách chiết axit nucleic không được liệt kê ở đây mà phụ thuộc vào phương pháp đã được chấp nhận (9.3).
5.3. Thuốc thử dùng cho PCR
Xem TCVN 7682 (ISO 20838).
5.3.1. Oligonucleotid (mồi) và mẫu dò phát hiện
Các mồi và mẫu dò dùng để phát hiện các trình tự gen đích đặc hiệu bằng PCR chuẩn và real-time PCR được liệt kê trong Phụ lục C và Phụ lục E.
6. Thiết bị, dụng cụ
Chỉ sử dụng các thiết bị, dụng cụ phòng thử nghiệm vi sinh [xem TCVN 6404 (ISO 7218)] và cụ thể như sau:
6.1. Nồi cách thủy hoặc khối ổn nhiệt, có thể duy trì ở nhiệt độ đến 100 oC.
6.2. Tủ ấm, theo TCVN 6404 (ISO 7218), có thể duy trì ở nhiệt độ 37 oC ± 1 oC.
6.3. Thiết bị tách chiết axit nucleic
Thiết bị thích hợp phụ thuộc vào phương pháp được chấp nhận (nếu cần).
6.4. Pipet, dung tích từ 1 μl đến 100 μl, phù hợp vớ ISO 7550.[16]
6.5. Ống nghiệm nhỏ real-time PCR có thành mỏng (0,2 ml/0,5 ml ống phản ứng), khay vi thể PCR nhiều giếng hoặc dụng cụ bằng chất dẻo trong suốt thích hợp khác dùng một lần.
6.6. Chu trình nhiệt. Một số loại thiết bị có sẵn và có thể lựa chọn theo nguyên tắc của phòng thử nghiệm.
6.7. Thiết bị phát hiện sản phẩm PCR
Phát xạ ánh sáng sau phép thử PCR 5’ nuclease bằng thiết bị real-time PCR.
6.8. Bộ trộn kiểu nhu động, có các túi vô trùng, có thiết bị điều chỉnh tốc độ và thời gian.
7. Lấy mẫu
Việc lấy mẫu không quy định trong tiêu chuẩn này. Xem các tiêu chuẩn cụ thể đối với sản phẩm có liên quan hoặc các quy định cụ thể. Nếu không, các bên có liên quan cần thỏa thuận về vấn đề này.
Mẫu gửi đến phòng thử nghiệm phải là mẫu đại diện. Mẫu không bị hư hỏng trong suốt quá trình bảo quản hoặc vận chuyển.
8. Chuẩn bị mẫu thử
Chuẩn bị mẫu thử theo tiêu chuẩn cụ thể đối với sản phẩm có liên quan. Nếu không, các bên có liên quan cần thỏa thuận về vấn đề này.
9. Cách tiến hành
9.1. Phần mẫu thử và huyền phù ban đầu
9.1.1. Yêu cầu chung
Sử dụng lượng môi trường tăng sinh cần thiết để thu được dung dịch pha loãng cuối cùng 10-1 của phần mẫu thử ban đầu.
9.1.2. Đối với các nền mẫu giả định có hệ vi sinh vật nền ở mức cao
Đối với các nền mẫu dạng rắn, chuyển phần mẫu thử (x g) trong điều kiện vô trùng vào túi của bộ trộn nhu động có chứa sẵn 9x ml mTSB đã được bổ sung novobiocin hoặc acriflavin (5.1.1.4). Tốt nhất là dùng các túi có lọc.
Đồng hóa mẫu trong bộ trộn nhu động [xem TCVN 6404 (ISO 7218)] (6.8).
Đối với các nền mẫu dạng lỏng, dùng pipet vô trùng chuyển phần mẫu thử (x ml) vào ống hoặc lọ có chứa sẵn 9x ml mTSB tăng sinh đã được bổ sung novobiocin hoặc acriflavin (5.1.1.4).
9.1.3. Đối với các nền mẫu giả định có vi khuẩn đích bị ức chế
Để các sản phẩm đông lạnh rã đông ở nhiệt độ phòng, sau đó chuyển phần mẫu thử (x g hoặc x ml)
sang túi của bộ trộn nhu động hoặc ống có chứa sẵn 9x ml BPW (5.1.2) và tiến hành như trên.
9.2. Tăng sinh
9.2.1. Ủ
Ủ túi của bộ trộn nhu động, ống hoặc lọ (9.1.2) ở 37 oC ± 1 oC trong 18 h đến 24 h.
9.2.2. Kiểm soát quá trình (đối với real-time PCR)
Thực hiện kiểm soát quá trình theo ISO 22174.
Hướng dẫn về việc kiểm soát khuếch đại bên trong (IAC) và kiểm soát quá trình nêu trong Phụ lục D và C.3.8.
9.3. Tách chiết axit nucleic
Sử dụng quy trình tách chiết axit nucleic thích hợp đối với vi khuẩn Gram âm. Tất cả các phương pháp có trong Tài liệu tham khảo [10]. Ngoài ra, có thể sử dụng các bộ kit thương mại theo hướng dẫn của nhà sản xuất.
9.4. Khuếch đại PCR (đối với real-time PCR)
9.4.1. Yêu cầu chung
Phương thức khuếch đại PCR được quy định dựa trên khuếch đại real-time PCR.
Thực hiện tất cả các yêu cầu đối với phương thức khuếch đại PCR theo quy định trong TCVN 7682 (ISO 20838).
Các mồi và các mẫu dò phát hiện đối với real-time PCR được quy định trong Phụ lục E.
9.4.2. Phát hiện các sản phẩm PCR
Ánh sáng phát xạ ngay khi sinh ra trong quá trình khuếch đại được bắt giữ bằng thiết bị.
9.4.3. Diễn giải các kết quả PCR
Các kết quả PCR thu được, bao gồm cả các kiểm soát quy định trong ISO 22174 và trong Phụ lục D được giải thích bằng phần mềm kết nối với thiết bị. Trong suốt quá trình khuếch đại, phần mềm giám sát quá trình khuếch đại PCR 5’ nuclease bằng phân tích phát xạ huỳnh quang của chất màu chỉ thị đối với từng mẫu, Rn. ΔRn là Rn trừ đi cường độ màu của đường nền được thiết lập trong vài chu trình đầu tiên. Vào cuối chu trình PCR, phản ứng được coi là dương tính, nếu đường ΔRn vượt quá ngưỡng, được xác định bằng 10 lần độ lệch chuẩn của phát xạ đường nền trung bình tính được giữa vài chu trình đầu tiên. Ngưỡng chu trình, Ct, được xác định là số chu trình mà tại đó giá trị huỳnh quang ΔRn của mẫu vượt quá giá trị ngưỡng xác định được.
Nếu các giá trị không rõ ràng, thì kiểm tra lại đường phát xạ. Các mẫu dương tính cho đồ thị tăng rõ về sự phát huỳnh quang, bắt đầu từ một số chu trình tương ứng với Ct.
Nếu các kiểm soát cho kết quả không như dự đoán thì lặp lại quy trình.
Phương pháp này được thực hiện theo các bước liên tiếp (xem sơ đồ trong Hình A.1) như sau:
– bước 1: phát hiện các gen mã hóa Stx và gen eae (phương pháp PCR A trong Phụ lục E – bước này cũng có thể được thực hiện bằng phương pháp PCR kép).
– bước 2 a): các mẫu dương tính đối với các gen stx và gen eae được kiểm tra về nhóm huyết thanh phân tử (phương pháp PCR B trong Phụ lục E);
– bước 2 b): các mẫu dương tính đối với các gen mã hóa Stx được phân lập đến chủng – phương pháp tăng sinh đặc hiệu nhóm huyết thanh (ví dụ: IMS) có thể được sử dụng để cải thiện quá trình phân lập STEC từ các mẫu dương tính với một trong các nhóm huyết thanh nằm trong phạm vi áp dụng của tiêu chuẩn này (xem 9.5 và Hình B.1).
9.5. Phân lập chủng
Cần phân lập các chủng STEC để khẳng định rằng các tín hiệu PCR dương tính được tạo ra từ các gen có mặt trong cùng tế bào vi khuẩn sống.
Trong trường hợp dương tính với một trong các gen liên quan đến các nhóm huyết thanh nêu trong phạm vi áp dụng của tiêu chuẩn này, thì có thể dùng phương pháp tăng sinh đặc hiệu nhóm huyết thanh để tạo thuận lợi cho bước phân lập bằng cách cấy trực tiếp vi sinh vật trên môi trường rắn thích hợp và sàng lọc các khuẩn lạc có gen độc.
Các quy trình PCR chuẩn và real-time PCR nêu trong Phụ lục C hoặc Phụ lục E hoặc sử dụng quy trình bất kỳ khác tương đương với quy trình PCR để khẳng định sự có mặt của các gen độc trong các khuẩn lạc phân lập được.
Sơ đồ quy trình phân lập STEC được nêu trong Hình B.1 và quy trình phân lập được nêu trong Phụ lục F.
10. Biểu thị kết quả
a) các mẫu âm tính đối với các gen stx: STEC không phát hiện có trong phần mẫu thử x g hoặc x ml [xem TCVN 6404 (ISO 7218)].
Khi không có mặt gen stx thì dừng quy trình không xác định tiếp các gen eae mã hóa intimin hoặc các gen liên quan đến các nhóm huyết thanh trong phạm vi áp dụng của tiêu chuẩn này.
Nếu không thực hiện được quá trình phân lập từ các mẫu dương tính để sàng lọc gen stx thì:
b) các mẫu dương tính với gen stx: phát hiện STEC giả định trong phần mẫu thử x g hoặc x ml.
c) các mẫu dương tính với gen stx và gen eae: phát hiện STEC giả định gây kết dính và gây phân hủy trong phần mẫu thử x g hoặc x ml.
d) các mẫu dương tính với gen stx và gen eae cũng như các gen có liên quan đến một trong các nhóm huyết thanh nêu trong phạm vi áp dụng của tiêu chuẩn này: phát hiện giả định STEC của nhóm huyết thanh XX1) trong phần mẫu thử x g hoặc x ml.
Nếu thực hiện được quá trình phân lập và khẳng định từ các mẫu dương tính để sàng lọc gen stx:
e) các chủng E. coli phân lập dương tính với gen stx: có mặt STEC trong phần mẫu thử x g hoặc x ml.
f) các chủng E. coli phân lập dương tính với gen stx và gen eae: có mặt STEC gây kết dính và gây phân hủy trong phần mẫu thử x g hoặc x ml
g) các chủng E. coli phân lập được dương tính với các gen stx và gen eae cũng như các gen có liên quan đến một trong các nhóm huyết thanh nêu trong phạm vi áp dụng của tiêu chuẩn này: có mặt STEC của nhóm huyết thanh XX2) trong phần mẫu thử x g hoặc x ml.
11. Dữ liệu về hiệu năng
11.1. Phương pháp real-time PCR nêu trong tiêu chuẩn này đã được đánh giá xác nhận theo ISO 16140:2003[17]. Vì tạm thời chỉ có TCVN 7686 (ISO 16654)[19] được dùng làm phương pháp chuẩn để phát hiện E. coli O157 trong thực phẩm nên phương pháp này chỉ được NF đánh giá, xác nhận và chứng nhận (Tài liệu tham khảo [7]) để phát hiện chỉ STEC thuộc nhóm huyết thanh O157.
Tiếp theo nghiên cứu này, một phần của phương pháp có liên quan đến STEC O157 đã được AFNOR chứng nhận là tương đương với tiêu chuẩn ISO 16140:2003 (chứng nhận số GEN 25/04-11/08) Hồ sơ đánh giá xác nhận hoàn chỉnh có sẵn trong Tài liệu tham khảo [7].
11.2. Một số đặc tính về hiệu năng giai đoạn sàng lọc real-time PCR của phương pháp đã được xác định trong các nghiên cứu đã được công bố. Cụ thể, độ nhạy và giới hạn phát hiện của phương pháp real-time PCR được xác định bằng cách sử dụng các dịch pha loãng của các plasmid có chứa gen dòng vô tính mã hóa các gen đích khác nhau (Tài liệu tham khảo [8]). Kết quả của nghiên cứu này được nêu trong Bảng 1.
Bảng 1 – Độ nhạy và giới hạn phát hiện của phép phân tích PCR 5’ nuclease đối với một số gen đích nêu trong tiêu chuẩn này (Tài liệu tham khảo [8])
|
Gen đích |
Giới hạn phát hiện Số lần sao chép/phản ứng |
Hiệu suất % |
|
stx1 |
5 |
94,7 |
|
stx2 |
5 |
94 |
|
rfbE |
1 |
94,2 |
|
wbdl |
5 |
94 |
|
wzx |
5 |
97,7 |
|
ihp1 |
5 |
99,6 |
Một nghiên cứu khác (Tài liệu tham khảo [17]) đã xem xét hiệu năng của các phương pháp tiếp cận real-time PCR trong quá trình sàng lọc hỗn hợp chủng cấy STEC thuộc các nhóm huyết thanh trong phạm vi áp dụng của tiêu chuẩn này, với chủng phòng thí nghiệm K-12 (C600). Các kết quả được nêu trong Bảng 2.
Bảng 2 – Phát hiện số lượng STEC thấp trong môi trường hỗn hợp (phù hợp với Tài liệu tham khảo [15])
|
Kiểu huyết thanh STEC |
CFU/ml |
Giá trị Ct |
|||||||
|
stx1/stx2 |
eae |
rfbE (O157) |
wbd1 (O111) |
wzx (O103) |
ihp1 (O145) |
fliC (H7) |
wzx (O26) |
||
|
O26:H11 |
2 đến 3 |
28 đến 31 |
31 đến 32 |
— |
— |
— |
— |
— |
31 đến 33 |
|
|
|
|
|
|
|
|
|
|
|
|
10 đến 20 |
25 đến 27 |
28 đến 29 |
— |
— |
— |
— |
— |
29 |
|
|
O103:H2 |
2 đến 3 |
29 đến 30 |
31 đến 32 |
— |
— |
32 đến 33 |
— |
— |
— |
|
|
|
|
|
|
|
|
|
|
|
|
10 đến 20 |
26 đến 27 |
29 đến 30 |
— |
— |
29 đến 31 |
— |
— |
— |
|
|
O111:[H8] |
2 đến 3 |
26 đến 27 |
30 đến 31 |
— |
30 đến 31 |
— |
— |
— |
— |
|
|
|
|
|
|
|
|
|
|
|
|
10 đến 20 |
24 đến 25 |
29 đến 30 |
_ |
27 đến 28 |
— |
— |
— |
— |
|
|
O145:[H28] |
2 đến 3 |
32 đến 33 |
31 đến 32 |
— |
|
_ |
31 đến 34 |
— |
— |
|
|
|
|
|
|
|
|
|
|
|
|
10 đến 20 |
30 đến 32 |
29 đến 31 |
— |
— |
— |
30 |
— |
— |
|
|
O157:H7 |
2 đến 3 |
29 đến 30 |
29 đến 31 |
31 đến 32 |
— |
— |
— |
33 đến 38 |
— |
|
|
|
|
|
|
|
|
|
. |
|
|
10 đến 20 |
26 đến 28 |
26 đến 29 |
29 đến 31 |
— |
— |
— |
31 đến 33 |
— |
|
11.3. Tiêu chuẩn này được sử dụng trong nghiên cứu cộng tác lần thứ ba do Phòng thí nghiệm chuẩn của Liên minh Châu Âu (EURL) tổ chức năm 2009 về E. coli bao gồm cả STEC. Nghiên cứu này bao gồm việc kiểm tra tập hợp năm mẫu gạc bông mô phỏng (bọt biển đã được làm ẩm) có chứa các vi sinh vật cần nghiên cứu, bao gồm cả STEC O157 và STEC O26, cùng với hệ vi sinh vật nền (Bảng 3). Chọn các mẫu gạc bông mô phỏng làm nền mẫu để phân tích trong các phép thử thành thạo theo nguyên tắc có trong các hướng dẫn của Cơ quan An toàn Thực phẩm Châu Âu cho các kế hoạch thử nghiệm tiếp theo đối với STEC [TCVN 7686 (ISO 16654)][19].
Có 14 phòng thử nghiệm đạt chuẩn quốc gia (NRL) về E. coli tham gia nghiên cứu. Việc đánh giá hiệu quả của phương pháp đối với STEC không phải O157 (STEC O26) cho các giá trị như sau:
Độ nhạy (Se): 100 % [95 % trong khoảng tin cậy (Cl) từ 96,97 % đến 100 %];
Độ nhạy (Sp): 99,62 % [95 % trong khoảng tin cậy (Cl) từ 97,5 % đến 100 %].
Các kết quả phân tích của mỗi NRL được nêu trong Bảng 4.
Các NRL cũng được yêu cầu phân lập STEC không phải O157 có trong các mẫu. Kết quả của bước phân lập nêu trong Bảng 5.
Báo cáo các kết quả phân tích đầy đủ của nghiên cứu cộng tác EU-RL-STEC lần thứ 3 được công khai rộng rãi.[9]
Bảng 3 – Thành phần mẫu trong nghiên cứu cộng tác EURL-STEC lần thứ 3
Các giá trị tính bằng đơn vị hình thành khuẩn lạc trên mililít
|
Mẫu/chất nhiễm bẩn |
Mẫu A |
Mẫu B |
Mẫu C |
Mẫu D |
Mẫu E |
|
STEC O157 stx1, stx2, eae |
2 |
2 x 103 |
20 |
0 |
0 |
|
STEC O26 stx1, eae |
0 |
40 |
4 x 103 |
40 |
0 |
|
E. coli |
102 |
102 |
102 |
102 |
102 |
|
K. pneumoniae |
2 x 102 |
2 x 102 |
2 x 102 |
2 x 102 |
2 x 102 |
|
S. faecalis |
5 x 102 |
5 x 102 |
5 x 102 |
5 x 102 |
5 x 102 |
Độ không đảm bảo đo mở rộng, U, có liên quan với mức chủng cấy là 0,22 log10(CFU/ml) đối với các chủng E. coli xác định được theo TCVN 9332:2012 (ISO/TS 19036:2006).[20]
Bảng 4 – Phát hiện các gen độc và các gen có liên quan đến nhóm huyết thanh trong các mẫu được tăng sinh bằng phương pháp real-time PCR
(phần sàng lọc của phương pháp; nghiên cứu cộng tác EURL-STEC lần thứ ba)
|
Gen đích |
Mẫu |
Phòng thử nghiệm tham gia |
||||||||||||||
|
L1 |
L2 |
L4 |
L7 |
L8 |
L9 |
L12 |
L14 |
L15 |
L17 |
L21 |
L22 |
L25 |
L30 |
|||
|
stxa |
A |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
|
B |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
|
|
C |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
|
|
D |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
|
|
E |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
|
|
eae |
A |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
|
B |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
|
|
C |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
|
|
D |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
|
|
E |
– |
– |
– |
– |
– |
– |
– |
– |
– |
+ |
– |
– |
– |
– |
– |
|
|
O26 |
A |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
|
B |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
|
|
C |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
|
|
D |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
|
|
E |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
|
|
O111 |
A |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
|
B |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
|
|
C |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
|
|
D |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
|
|
E |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
|
|
O103 |
A |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
|
B |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
|
|
C |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
|
|
D |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
|
|
E |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
|
|
O145 |
A |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
|
B |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
|
|
C |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
|
|
D |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
|
|
E |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
– |
|
|
a Bảng này bao gồm cả stx1 hoặc stx2. |
||||||||||||||||
Bảng 5 – Phân lập STEC O26 từ các mẫu được tăng sinh dương tính với RT PCR
(Khẳng định các dịch cấy dương tính bằng cách phân lập các phần có chủng bị ảnh hưởng, nghiên cứu cộng tác EURL-STEC lần thứ ba)
|
Phép thử |
Mẫu |
Giá trị đúng |
Phòng thử nghiệm tham gia |
|||||||||||||
|
L1 |
L2 |
L4 |
L7 |
L8 |
L9 |
L12 |
L14 |
L15 |
L17 |
L21 |
L22 |
L25 |
L30 |
|||
|
Phân lập O26 |
B |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
|
C |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
|
|
D |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
+ |
|
PHỤ LỤC A
(Quy định)
Sơ đồ quy trình sàng lọc
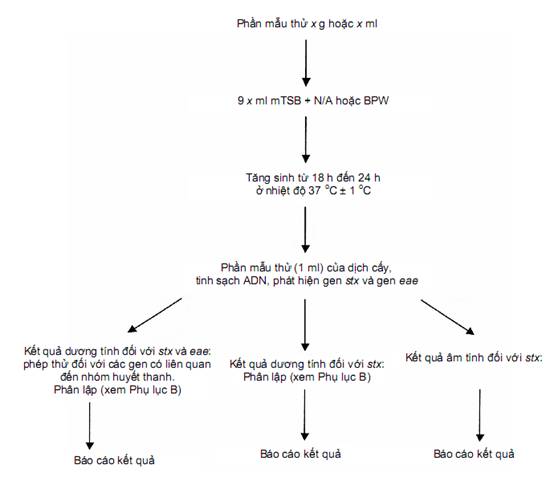
Hình A.1 – Sơ đồ quy trình sàng lọc
PHỤ LỤC B
(Quy định)
Sơ đồ quy trình phân lập và khẳng định3)
Nếu mẫu là dương tính đối với một trong các gen có liên quan đến nhóm huyết thanh nêu trong phạm vi áp dụng của tiêu chuẩn này thì có thể tăng sinh một nhóm huyết thanh đặc hiệu (SSE) để tạo thuận tiện cho quá trình phân lập.
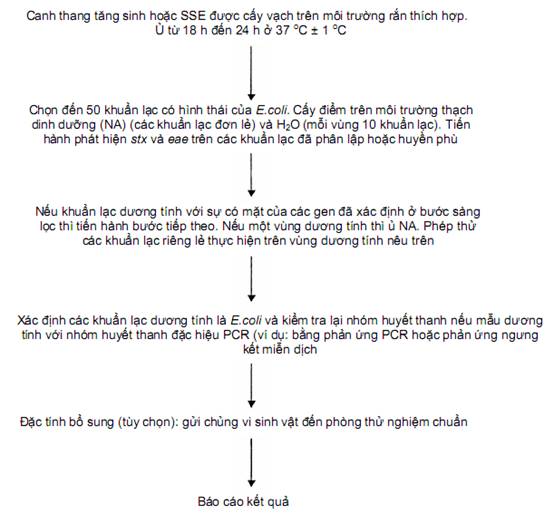
Hình B.1 – Sơ đồ quy trình phân lập và khẳng định
PHỤ LỤC C
(Tham khảo)
Nhận biết Escherichia coli sinh độc tố Shiga (STEC) bằng phương pháp khuếch đại PCR đa mồi các gen độc và phát hiện các sản phẩm của phản ứng PCR bằng điện di trên gel agarose
C.1. Yêu cầu chung
STEC là các chủng Escherichia coli có chứa thể thực khuẩn phân giải mang các gen mã hóa sinh độc tố Shiga (Tài liệu tham khảo [12]). Thực hiện phương pháp quy định trong Phụ lục này để phát hiện sự có mặt các gen mã hóa Stx trong chủng cấy E. coli bằng PCR đa mồi, để nhận biết chúng là STEC. Việc xác định sự có mặt các gen eae mã hóa intimin cũng bao gồm trong phụ lục này vì nó liên quan đến các chủng STEC gây bệnh ở người.
Sử dụng các cặp mồi stx1Flstx1R và stx2Flstx2R (Tài liệu tham khảo [11]), có thể phát hiện được các gen stx1 và stx2, tương ứng. Gen stx2 xác nhận tất cả các biến thể của stx2, trừ stx2f. Tuy nhiên, biến thể này không được coi là ảnh hưởng được đến sức khỏe cộng đồng, mà phần lớn quan sát được trong STEC được phân lập từ các loài chim (ISO/TS 19036[20]). Các mồi được sử dụng để phát hiện eae (Tài liệu viện dẫn [11]) xác nhận tất cả các biến thể đa hình ghi được của gen này.
CẢNH BÁO – Phương pháp nêu trong phụ lục này sử dụng các dịch cấy vi khuẩn thuần khiết chỉ để khẳng định các chủng phân lập được.
C.2. Chữ viết tắt
S.U.: Đơn vị mẫu.
C.3 Cách tiến hành
C.3.1 Nguyên tắc của phương pháp
Phương pháp này dựa trên sự khuếch đại các vùng ADN đặc hiệu bằng PCR từ một khuôn mẫu ADN, với các oligonucleotid khởi động bước đầu phản ứng PCR.
Việc phát hiện các gen stx1, stx2 và eae được thực hiện bằng phản ứng PCR đa mồi sử dụng các mồi đặc hiệu (Bảng C.1). Phương pháp này bao gồm các bước dưới đây:
– chuẩn bị khuôn mẫu;
– chuẩn bị phản ứng PCR;
– xác định các kết quả PCR bằng điện di trên gel agarose.
C.3.2 Chuẩn bị khuôn mẫu
Dịch nuôi cấy được cấy vạch trên môi trường rắn, ví dụ: thạch trypton-đậu tương (TSA), được thực hiện như sau:
– dùng que cấy vòng vô trùng 1 μl lấy một khuẩn lạc đơn lẻ của vi khuẩn;
– chuẩn bị khuôn mẫu bằng cách tạo huyền phù vi khuẩn trong 100 μl nước milliQ4) đã lọc qua bộ lọc vô trùng cỡ lỗ 0,22 μm và đun sôi trong 10 min.
C.3.3. Chuẩn bị phản ứng PCR
Đối với mỗi mẫu thử, chuẩn bị 50 μl cho phản ứng [chất đệm phản ứng 1×, MgCl2 1,2 mmol/l, mỗi deoxynucleotidetriphosphat (dNTP) 0,2 mmol/l, mỗi mồi 50 pmol, 2 đơn vị Taq polymerase và 10 μl khuôn mẫu ADN]. Xác định thể tích thuốc thử theo thể tích cuối của phản ứng. Sử dụng nước milliQ cho các phản ứng PCR.
Trong mỗi phép phân tích PCR, gồm có một kiểm soát dương tính và hai kiểm soát âm tính. Kiểm soát dương tính là một khuôn mẫu ADN thu được từ một chủng E. coli có các gen độc cần thử nghiệm, một kiểm soát âm tính là ADN từ chủng E. coli không gây bệnh (không có các gen độc tiềm ẩn) và một kiểm soát âm tính còn lại là từ khuôn mẫu không có ADN mẫu thử.
Giữ các mẫu phản ứng trong chu trình nhiệt đã được cài đặt theo chương trình nhiệt nêu trong Tài liệu tham khảo [11] (Bảng C.1).
C.3.4. Điện di trên gel agarose
Chuẩn bị 20 g/l gel agarose trong 1× tris/borat/EDTA (TBE) hoặc tris/axetat/EDTA (TAE). Cho vào mỗi giếng gel với 15 μl mỗi loại thuốc thử hỗn hợp PCR với chất nhuộm ở nồng độ cuối 1×. Cho chạy các mẫu chất đệm 1× (TBE hoặc TAE) ở điện áp không đổi (100 V). Sử dụng chất đánh dấu có khối lượng phân tử phù hợp với khối lượng phân tử chính xác cần khuếch đại (xem Bảng C.1).
CẢNH BÁO – Cần lưu ý rằng sự phân bố chính xác dải băng là một điểm rất quan trọng trong việc đánh giá sự có mặt của các gen độc. Cần đảm bảo các dải băng được tạo ra bởi các chủng đối chứng phù hợp chính xác với khối lượng phân tử dự kiến.
Ethidi bromua cần được bổ sung vào gel agarose để hiển thị ADN. Thuốc thử này là chất xen giữa gây đột biến ADN thường được sử dụng làm chất nhuộm màu axit nucleic trong các phòng thí nghiệm sinh học phân tử. Khi tiếp xúc với ánh sáng UV, thuốc thử phát huỳnh quang có màu đỏ-da cam. Trước khi rót gel agarose vào khuôn gel điện di, nồng độ cuối của ethidi bromua được bổ sung phải là 0,5 μg/ml. Ngoài ra, gel agarose có thể được nhuộm màu sau khi điện di trong dung dịch ethidi bromua 0,5 μg/ml.
Dung dịch nhuộm gel khác với ethidi bromua là có sẵn và được sử dụng theo hướng dẫn của nhà sản xuất.
C.3.5. Thiết bị, dụng cụ
Chỉ sử dụng các thiết bị, dụng cụ phòng thử nghiệm vi sinh [xem TCVN 6404 (ISO 7218)] và cụ thể như sau:
C.3.5.1. Tủ hút vô trùng, loại dùng cho PCR.
C.3.5.2. Pipet vô trùng, dung tích 1 ml.
C.3.5.3. Vòng cấy vô trùng, dùng cho vi khuẩn.
C.3.5.4. Lọ thủy tinh borosilicat, dung tích 500 ml.
C.3.5.5. Xyranh thủy tinh borosilicat, dung tích 500 ml.
C.3.5.6. Xyranh thủy tinh borosilicat, dung tích 1 lít.
C.3.5.7. Tủ ấm, có thể duy trì nhiệt độ ở 37 oC ± 1 oC.
C.3.5.8. Cân bàn kỹ thuật.
C.3.5.9. Nồi hấp áp lực.
C.3.5.10. Giá đỡ Pipet.
C.3.5.11. Micropipet.
C.3.5.12. Đầu tip micropipet, vô trùng.
C.3.5.13. Ống li tâm nhỏ, dung tích 1,5 ml.
C.3.5.14. Ống PCR, dung tích 0,2 ml hoặc 0,5 ml.
C.3.5.15. Chu trình nhiệt.
C.3.4.16. Máy khuấy từ.
C.3.5.17. Que khuấy từ.
C.3.5.18. Máy khử ion miliQ.
C.3.5.19. Thiết bị điện di.
C.3.5.20. Máy rọi UV.
C.3.5.21. Máy làm đá lạnh.
C.3.5.22. Lò vi sóng.
C.3.6. Thuốc thử và môi trường
Chỉ sử dụng các thuốc thử loại tinh khiết phân tích và nước cất hoặc nước đã khử khoáng hoặc nước có độ tinh khiết tương dương, trừ khi có quy định khác.
C.3.6.1. Đĩa thạch.
C.3.6.2. Dung dịch gốc dNTP.
C.3.6.3. Dung dịch oligonucleotid tổng hợp.
C.3.6.4. Polymerase ADN Taq và dung dịch đệm phản ứng 10× có hoặc không có MgCl2.
C.3.6.5. Dung dịch đệm chạy điện di.
C.3.6.6. Chất đánh dấu khối lượng phân tử ADN.
C.3.6.7. Chất nhuộm màu.
C.3.6.8. Agarose
C.3.6.9. Dung dịch ethidi bromua hoặc chất nhuộm gel ADN khác.
C.3.7. An toàn và thiết bị bảo vệ
Một số chủng STEC có thể lây nhiễm sang người với liều lượng rất thấp và có thể gây bệnh nghiêm trọng. Các bệnh truyền nhiễm bị mắc từ phòng thử nghiệm phải được báo cáo. Vì vậy thao tác với STEC cần phải có thực hành phòng thử nghiệm tốt và sử dụng các thiết bị bảo vệ. Ethidi bromua là chất gây đột biến gen và là chất độc, vì vậy cần sử dụng các tấm bảo vệ cùng với các thiết bị bảo vệ phù hợp (áo choàng phòng thử nghiệm và găng tay cao su). Ánh sáng UV có thể gây hại cho mắt vì vậy việc sử dụng các màn chắn polymetylmeacrylat và kính bảo hộ là bắt buộc.
C.3.8. Các chủng đối chứng và kiểm soát quá trình
Sử dụng chủng STEC có chứa các gen stx1, stx2 và eae làm chủng kiểm soát dương tính đối với tất cả các gen này. Ví dụ: chủng đối chứng E. coli O157 EDL933 (WDCM 00188) (Tài liệu tham khảo [12][13]).
Sử dụng mọi chủng E. coli K12, cũng như MG1655 làm chủng kiểm soát âm tính.
Việc kiểm soát phương pháp PCR được thực hiện theo quy định trong C.3.2. Mẫu kiểm soát có thể được chuẩn bị trước và được bảo quản trong 10 μl dung dịch chuẩn bị sẵn, mẫu này dùng được trong 8 tháng ở − 20 oC.
C.3.9. Diễn giải các kết quả
Các mẫu cho các phân đoạn khuếch đại có kích thước dự kiến (xem C.3.4 và Bảng C.1) được coi là dương tính đối với các gen đích có liên quan.
Trong mỗi phản ứng, bao gồm cả các mẫu kiểm soát dương tính và kiểm soát âm tính, cho các kết quả dương tính và âm tính tương ứng. Nếu các mẫu kiểm soát cho các kết quả không chính xác thì lặp lại toàn bộ quy trình.
Đánh giá, xác nhận sản phẩm khuếch đại bằng trình tự trực tiếp hoặc bằng các phương pháp thích hợp khác có thể là không cần thiết vì phương pháp này dùng để khẳng định các chủng phân lập được và phụ thuộc vào phương pháp real-time PCR đối với các đặc tính tương tự.
Việc xác nhận các sản phẩm khuếch đại (ví dụ: bằng trình tự trực tiếp hoặc bằng cách phân tích endonuclease giới hạn) cần được thực hiện khi thu được các kết quả không rõ ràng.
Bảng C.1 – Các phân đoạn khuếch đại có kích thước dự kiến
|
Gen đích |
Tên mồi[11] |
Trình tự mồi |
Cỡ sản phẩm khuếch đại |
|
eae |
eaeAF |
GAC CCG GCA CAA GCA TAA GC |
384 |
|
eaeAR |
CCA CCT GCA GCA ACA AGA GG |
||
|
stx1 |
stx1F |
ATA AAT CGC CAT TCG TTG ACT AC |
180 |
|
stx1R |
AGA ACG CCC ACT GAG ATC ATC |
||
|
stx2 (nhóm) |
stx2F |
GGC ACT GTC TGA AAC TGC TCC |
255 |
|
stx2R |
TCG CCA GTT ATC TGA CAT TCTG |
||
|
Chu trình nhiệt[11]: 35 chu trình PCR, mỗi chu trình gồm có: giai đoạn biến tính trong 1 min ở 95 oC, giai đoạn bắt cặp trong 2 min ở 65 oC ở 10 chu trình đầu tiên, biến tính đến 60 oC từ chu trình 11 đến chu trình 15 (1 oC trên chu trình) và giai đoạn kéo dài trong 1,5 min ở 72 oC, tăng đến 2,5 min từ chu trình 25 đến chu trình 35. |
|||
Phụ lục D
(Tham khảo)
Kiểm soát độ khuếch đại bên trong
Có thể sử dụng ba phép kiểm soát độ khuếch đại bên trong phản ứng real-time PCR:
– TaqMan®5) là kiểm soát dương tính bên trong ngoại sinh. Bộ kit thuốc thử bao gồm tất cả các thuốc thử cần thiết (mồi, VicTM6), mẫu dò, ADN đích IAC và dung dịch ức chế). Cần pha loãng AND đích IAC 10 lần để thu được khoảng 100 bản sao trên một phản ứng PCR. Chiều dài sản phẩm PCR không được công khai với khách hàng.
– Đối với phép tính pUC 19 thông thường dựa vào kiểm soát độ khuếch đại bên trong IAC, xem Tài liệu tham khảo [27]. Nên sử dụng khoảng 100 bản sao ADN đích (pUC 19) trên phản ứng PCR. Cỡ IAC là 119 bp.
– Có thể sử dụng plasmid tái tổ hợp (được gọi là pIAC-STEC) trong thử nghiệm real-time PCR đối với gen stx đặc hiệu7). IAC này có chứa phân đoạn ADN đơn dòng vô tính trong vùng EcoRI của pUC 19 dưới đây:
5’-ATTTTTGTTACTGTGACAGCTGAAGCTTTACGTGAATCGCCAGCGGCATCAGCACCTTGTCGCCTTGCGTATAGATGTTGATCTTACATTGAACTGGGGAATT-3’ (các chữ in đậm: các mồi xuôi và mồi ngược của gen stx1/stx2 liên kết trình tự các vị trí liên tiếp; trình tự gạch chân: các vị trí liên kết IAC- mẫu dò).
IAC đồng khuếch đại với các gen stx sử dụng cùng các mồi là stx (Phụ lục E), dưới các điều kiện như nhau và trong cùng ống PCR.[14] IAC được phát hiện bằng các mẫu dò ADN đặc hiệu (5’ [Red640]- CAAGGCGACAAGGTGCTGATGCCG-[BHQ2] 3’ bao gồm cả trong hỗn hợp PCR ở nồng độ giống với nồng độ của các mẫu dò AND gen stx1 và gen stx2 (cả hai được đánh dấu bằng [floresxein] và BHQ1] tại vùng kết thúc 5’ và 3’ tương ứng). Cần sử dụng tổng số 64 bản sao IAC trên một phản ứng PCR. Các sản phẩm PCR của IAC dài 96 bp. Việc thực hiện phương pháp real-time PCR đối với gen stx-IAC cho thấy việc sử dụng các mẫu thực phẩm bị nhiễm bẩn tự nhiên hoặc nhân tạo (Tài liệu tham khảo [5]).
Cũng có thể sử dụng hệ các thống thứ hai và thứ ba làm kiểm soát quá trình chiết bằng cách bổ sung 100 bản sao plasmid pUC 19 hoặc pIAC-STEC vào mẫu dạng lỏng trước bước tinh sạch ADN.
PHỤ LỤC E
(Tham khảo)
Mồi và mẫu dò dùng cho phép phân tích PCR
Nguyên tắc real-time PCR được quy định dựa trên việc sử dụng các mồi và các mẫu dò dưới đây làm thuốc thử đối chứng. Tuy nhiên, các phương pháp khác bao gồm cả việc sử dụng các mồi và các mẫu dò khác với điều kiện các mồi và các mẫu dò đó đã được công nhận tương đương với các mồi và các mẫu dò nêu trong Bảng E.1 và E.2, phù hợp với các nguyên tắc nêu trong ISO 16140:2003[17].
E.1. Mồi và mẫu dò
Bảng E.1 và E.2 cung cấp các trình tự mồi và trình tự mẫu dò, tương ứng đối với:
– Việc phát hiện các gen stx và eae bằng phương pháp real-time PCR (PCA A);
– Việc phát hiện các gen có liên quan đến nhóm huyết thanh sử dụng phương pháp real-time PCR (PCR B).
Trong Bảng E.1 và Bảng E.2, các chất chỉ thị huỳnh quang và chất hấp thụ huỳnh quang không được nhận biết vì điều này phụ thuộc lớn vào các thiết bị real-time PCR có sẵn trong từng phòng thử nghiệm.
Bảng E.1 – Các mồi và mẫu dò thoái hóa được dùng cho phản ứng PCR 5’-nuclease
|
Gen đích |
Trình tự mồi xuôi, mồi ngược và mẫu dò (từ đầu 5’ đến đầu 3’)a |
Kích thước amplicon bp |
Vị trí trong trình tự |
Bổ sung vào Ngân hàng Gen Số |
|
stx1 Tài liệu tham khảo [3] |
TTT GTY ACT GTS ACA GCW GAA GCY TTA CG CCC CAG TTC ARW GTR AGR TCM ACR TC Mẫu dò-CTG GAT GAT CTC AGT GGG CGT TCT TAT GTAA |
131 |
878 đến 906 938 đến 1008 941 đến 971 |
M16625 |
|
stx2b Tài liệu tham khảo [3] |
TTT GTY ACT GTS ACA GCW GAA GCY TTA CG CCC CAG TTC ARW GTR AGR TCM ACR TC Mẫu dò -TCG TCA GGC ACT GTC TGA AAC TGC TCC |
128 |
785 đến 813 887 đến 912 838 đến 864 |
X07865 |
|
eae Tài liệu tham khảo [2] |
CAT TGA TCA GGA TTT TTC TGG TGA TA CTC ATG CGG AAA TAG CCG TTA Mẫu dò-ATA GTC TCG CCA GTA TTC GCC ACC AAT ACC |
102 |
899 đến 924 1000 đến 979 966 đến 936 |
Z11541 |
|
a Trong trình tự Y là (C,T), trình tự S là (C, G), trình tự W là (A,T), trình tự R là (A,G), trình tự M là (A, C). b Quá trình tổ hợp mồi/mẫu dò này nhận dạng tất cả các biến thể của gen stx2 trừ gen stx2f. |
||||
Bảng E.2 – Mồi và mẫu dò được dùng để khuếch đại các gen đặc hiệu kháng nguyên O trong phản ứng PCR 5’-nuclease
|
Gen đích (nhóm huyết thanh) |
Trình tự mồi xuôi, mồi ngược và mẫu dò (từ đầu 5’ đến đầu 3’)a |
Kích thước amplicon bp |
Vị trí trong trình tự |
Bổ sung vào Ngân hàng Gen Số |
|
rfbE (O157) Tài liệu tham khảo [3] |
TTT CAC ACT TAT TGG ATG GTC TCAA CGA TGA GTT TAT CTG CAA GGT GAT Mẫu dò-AGG ACC GCA GAG GAA AGA GAG GAA TTA AGG |
88 |
348 đến 372 412 đến 435 381 đến 410 |
AF163329 |
|
wbdl (O111) Tài liệu tham khảo [3] |
CGA GGC AAC ACA TTA TAT AGT GCT TT TTT TTG AAT AGT TAT GAA CAT CTT GTT TAGC Mẫu dò-TTG AAT CTC CCA GAT GAT CAA CAT CGT GAA |
146 |
3464 đến 3489 3579 đến 3609 3519 đến 3548 |
AF078736 |
|
wzx (O26) Tài liệu tham khảo [3] |
CGC GAC GGC AGA GAA AATT AGC AGG CTT TTA TAT TCT CCA ACT TT Mẫu dò-CCC CGT TAA ATC AAT ACT ATT TCA CGA GGT TGA |
135 |
5648 đến 5666 5757 đến 5782 5692 đến5724 |
AF529080 |
|
ihp1 (O145) Tài liệu tham khảo [3] |
CGA TAA TAT TTA CCC CAC CAG TAC AG GCC GCC GCA ATG CTT Mẫu dò-CCG CCA TTC AGA ATG CAC ACA ATA TCG |
132 |
1383 đến 1408 1500 đến 1514 1472 đến 1498 |
AF531429 |
|
wzx (O103) Tài liệu tham khảo [4] |
CAA GGT GAT TAC GAA AAT GCA TGT GAA AAA AGC ACC CCC GTA CTT AT Mẫu dò-CAT AGC CTG TTG TTT TAT |
99 |
4299 đến 4323 4397 đến 4375 4356 đến 4373 |
AY532664 |
PHỤ LỤC F
(Quy định)
Phân lập các chủng STEC
Áp dụng các quy trình quy định để phân lập các chủng STEC từ các mẫu dương tính bằng phương pháp real-time PCR.
a) Trong trường hợp dương tính với một trong các gen liên kết với các nhóm huyết thanh trong phạm vi áp dụng của tiêu chuẩn này, thì có thể tăng sinh nhóm huyết thanh đặc hiệu (SSE) trên dịch cấy tăng sinh còn lại để tạo điều kiện thuận tiện cho quá trình phân lập STEC (xem Chú thích 1).
b) Cấy vạch dịch cấy tăng sinh hoặc SSE trên TBX hoặc môi trường thích hợp khác (xem Chú thích 2). Ủ trong 18 h đến 24 h ở 37 oC ± 1 oC.
c) Lấy 50 khuẩn lạc có hình thái E. coli hoặc có hình dạng đặc trưng (xem Chú thích 5) và cấy điểm trên thạch dinh dưỡng (NA) (xem Chú thích 3) và nước (các khuẩn lạc có thể được khoanh vùng thành tổng số 10 khuẩn lạc mỗi vùng trong nước).
d) Tiến hành phát hiện các gen mã hóa stx trên các khuẩn lạc phân lập được hoặc trên các vùng nước (xem Chú thích 4).
e) Nếu vùng nước là dương tính, quay ngược lại NA và phân tích các khuẩn lạc đơn lẻ tạo thành nhóm dương tính để lựa chọn một khuẩn lạc dương tính đơn lẻ.
f) Nhận biết các khuẩn lạc là E. coli và khẳng định sự có mặt của gen eae và nhóm huyết thanh nếu được nhận biết trong bước sàng lọc (ở đây là: sử dụng phương pháp PCR B nêu trong Phụ lục E), xem Chú thích 5.
g) Các mẫu phân lập có thể được gửi đến các phòng thử nghiệm chuẩn để phân tích các đặc tính tiếp theo.
CHÚ THÍCH 1: Có thể thực hiện tăng sinh nhóm huyết thanh đặc hiệu bằng cách sử dụng các hệ thống thu nạp miễn dịch như IMS hoặc tương đương. Nhìn chung, cần tham khảo các hướng dẫn của nhà sản xuất.
CHÚ THÍCH 2: Đối với các mẫu dương tính O157, có thể các phương pháp nêu trong TCVN 7686 (ISO 16654)[19] hoặc các phương pháp khác được đánh giá, xác nhận theo ISO 16140-2[18] là thích hợp. Chủng E. coli O157 lên men đường sorbitol nhạy với telurit có trong môi trường CT SMAC được chỉ ra trong OSP 1664.[19] Vì vậy, sử dụng đĩa phân lập SMAC thứ hai không có các chất kháng sinh là thích hợp. Khi không có các khuẩn lạc âm tính với đường sorbitol trên đĩa thạch, thì nêu rõ phương pháp sàng lọc các khuẩn lạc dương tính với đường sorbitol.
Để phân lập chủng STEC O26 có thể dùng môi trường rắn khác (MacConkey) có chứa ramnose thay vì chứa lactose thương mại có bán sẵn (RMAC). Điều này rất hiệu quả trong việc phân biệt các chủng STEC O26 không lên men ramnose từ các vi khuẩn E. coli khác.
CHÚ THÍCH 3: Một số loại môi trường thạch dinh dưỡng thương mại có bán sẵn để dùng cho đĩa hoặc tự chuẩn bị từ bột khô. Môi trường thạch dinh dưỡng không chọn lọc (ở đây là: TSA) là thích hợp để duy trì các khuẩn lạc cho phân tích các đặc tính tiếp theo. Thạch enterohemolizin cũng có thể được sử dụng, loại thạch này có lợi thế phát hiện quá trình enterohemolizin phổ biến của STEC gây bệnh trên người.
CHÚ THÍCH 4: Phương pháp real-time PCR được quy định trong tiêu chuẩn này có thể được dùng để khẳng định sự có mặt các gen stx và gen eae trong các chủng phân lập được. Phương pháp PCR thông thường có thể được sử dụng làm phương án thay thế (Phụ lục C).
CHÚ THÍCH 5: Có thể khẳng định khuẩn lạc là E. coli bằng cách sử dụng nhiều kit thử sinh hóa thương mại hoặc bằng cách đánh giá quá trình sinh indol. Có thể khẳng định nhóm huyết thanh bằng phương pháp PCR hoặc bằng sự ngưng kết với kháng huyết thanh thương mại.
THƯ MỤC TÀI LIỆU THAM KHẢO
[1] Scientific Opinion of the Panel on Biological Hazards (BIOHAZ)-Monitoring of verotoxigenic Escherichia coli (VTEC) and identification of human pathogenic VTEC types (Question No EFSA- Q-2007-036). Eur. Food Saf. Author. J. 2007, (579), pp. 1-61
[2] NIELSEN, E.M., ANDERSEN, M.T. Detection and characterization of verocytotoxin-producing Escherichia coli by automated 5′ nuclease PCR assay. J. Clin. Microbiol. 2003, 41, pp. 2884-2893
[3] PERELLE, S., DILASSER, F., GROUT, J., FACH, P. Detection by 5′ nuclease PCR of Shiga-toxin producing Escherichia coli O26, O55, O91, O103, O111, O113, O145 and O157:H7, associated with the world’s most frequent clinical cases. Mol. Cell Probes 2004,18, pp. 185-192
[4] PERELLE, S., DILASSER, F., GROUT, J., FACH, P. Detection of Escherichia coli serogroup O103 by realtime polymerase chain reaction. J. Appl. Microbiol. 2005, 98, pp. 1162-1168
[5] VIMONT, A., DELIGNETTE-MULLER, M.L., VERNOZY-ROZAND, C. Supplementation of enrichment broths by novobiocin for detecting Shiga toxin-producing Escherichia coli from food: A controversial use. Lett. Appl. Microbiol. 2007, 44, pp. 326-331
6] UEMURA, R., SUEYOSHI, M., NAGAYOSHI, M., NAGATOMO, H. Antimicrobial susceptibilities of Shiga toxin-producing Escherichia coli isolates from pigs with edema disease in Japan. Microbiol. Immunol. 2003, 47, pp. 57-61
[7] GENESYSTEMS. Validation AFNOR des méthodes alternatives d’analyse: Application à la microbiologie alimentaire [AFNOR validation of alternative analytical methods: Application to food microbiology]. Quimper: Adria Développement. 56 p. Available (viewed 2012-10-17) at: http://www.afnor-validation.org/rapports-synthese/GENESYSTEMS/Synt%20GEN%2025-06%2011-08%20(fr).pdf
[8] KAGKLI, D.-M, WEBER, T.P., VAN DEN BULCKE, M., FOLLONI, S., TOZZOLI, R., MORABITO, S., ERMOLLI, M., GRIBALDO, L, VAN DEN EEDE, G. Application of the modular approach to an in-house validation study of real-time PCR methods for the detection and serogroup determination of verocytotoxigenic Escherichia coli. Appl. Environ. Microbiol. 2011, 77, pp. 6954-6963
[9] EUROPEAN UNION REFERENCE LABORATORY VTEC. Proficiency tests and ring trials on detection and typing methods. Rome: Istituto Superiore di Sanita. Available (viewed 2012-10-17) at: http://www.iss. it/vtec/neww/cont.php?id=147&lanq=2&tipo=15
[10] SAMBROOK, J., RUSSEL., D.W. Molecular cloning: A laboratory manual, 3rd edition, 3 vols. Cold Sprin Harbor, NY: Cold Spring Harbor Laboratory Press, 2001
[11] PATON, A.W., PATON, J.C. Detection and characterization of Shiga toxigenic Escherichia coli by using multiplex PCR assays for stx1, stx2, eaeA, enterohemorrhagic E. coli hlyA, rfbO111, and rfbO157. J. Clin. Microbiol. 1998, 36, pp. 598-602
[12] O’BRIEN, A.D., NEWLAND, J.W., MILLER, S.F., HOLMES, R.K., SMITH, H.W., FORMAL, S.B. Shiga-like toxin-converting phages from Escherichia coli strains that cause hemorrhagic colitis or infantile diarrhea. Science 1984, 226, pp. 694-696
[13] PERNA, NT., PLUNKETT, III, G., BURLAND, V., MAU, B., GLASNER, J.D., ROSE, D.J., et al. Genome sequence of enterohaemorrhagic Escherichia coli O157:H7. Nature 2001, 409, pp. 529-533
[14] HOORFAR, J., MALORNY, B., ABDULMAWJOOD, A., COOK, N., WAGNER, M., FACH, P. Practical considerations in design of internal amplification controls for diagnostic PCR assays. J. Clin. Microbiol. 2004, 42, pp. 1863-1868
[15] BEUTIN, L, JAHN, S., FACH, P. Evaluation of the ‘GeneDisc’ real-time PCR system for detection of enterohaemorrhagic Escherichia coli (EHEC) O26, O103, O111, O145 and O157 strains according to their virulence markers and their O-and H-antigen-associated genes. J. Appl. Microbiol. 2009, 106, pp. 1122-32
[16] ISO 7550, Laboratory glassware – Disposable micropipettes
[17] ISO 16140:2003, Microbiology of food and animal feeding stuffs – Protocol for the validation of alternative methods
[18] ISO 16140-28), Microbiology of food and animal feeding stuffs – Method validation – Part 2: Protocol for the validation of proprietary methods against a reference method
[19] TCVN 7686 (ISO 16654), Vi sinh vật trong thực phẩm và thức ăn chăn nuôi – Phương pháp phát hiện Escherichia coli O157
[20] TCVN 9332:2012 (ISO/TS 19036:2006, With Amd. 1:2009), Vi sinh vật thực phẩm và thức ăn chăn nuôi – Hướng dẫn ước lượng độ không đảm bảo đo đối với các phép phân tích định lượng
[21] ISO 20837, Microbiology of food and animal feeding stuffs – Polymerase chain reaction (PCR) for the detection of food-borne pathogens – Requirements for sample preparation for qualitative detection
[22] SCHEUTZ, F., TEEL, L.D., BEUTIN, L, PlERARD, D., BUVENS, G., kARCH, H., MELLMANN, A., CAPRIOU, A., TOZZOLI, R., MORABITO, S., STROCKBINE, N.A., MELTON-CELSA, A.R., SANCHEZ, M., PERSSON, S., O’BRIEN, A.D. A multi-center evaluation of a sequence-based protocol to subtype Shiga toxins and standardize Stx nomenclature. J. Clin. Microbiol. 2012 Jul 3
[23] NATIONAL CENTER FOR EMERGING AND ZOONOTIC INFECTIOUS DISEASES. DIVISION OF FOODBORNE, WATERBORNE, AND ENVIRONMENTAL DISEASES. National Enteric Disease Surveillance: Shiga toxinn producing Escherichia coli (STEC) Annual Summary, 2009. Bethesda, MD: CDC, 2012
[24] EUROPEAN FOOD SAFETY AUTHORITY AND EUROPEAN CENTRE FOR DISEASE PREVENTION AND CONTROL. The European Union Summary Report on Trends and Sources of Zoonoses, Zoonotic Agents and Food-borne Outbreaks in 2010. Eur. Food Saf. Author. J. 2012, 10, p. 2597
[25] EUROPEAN FOOD SAFETY AUTHORITY. Technical specifications for the monitoring and reporting of verotoxigenic Escherichia coli (VTEC) on animals and food (VTEC surveys on animals and food). Eur. Food Saf. Author. J. 2009, 7, p. 1366
[26] SCHMIDT, H., SCHEEF, J., MORABITO, S., CAPRIOLI, A., WIELER, L.H., KARCH, H. A new Shiga toxin 2 variant (Stx2f) from Escherichia coli isolated from pigeons. Appl. Environ. Microbiol. 2000, 66, pp. 1205-8.
[27] FRICKER, M., MESSELHUSSER, U., BUSCH, U., SCHERER, S., EHLING-SCHULZ, M. Diagnostic real- time PCR assays for the detection of emetic Bacillus cereus strains in foods and recent food-borne outbreaks. Appl. Environ. Microbiol. 2007, 73, pp. 1892-1898
1) XX cho thấy nhóm huyết thanh được xác định bởi sự có mặt của các gen đang nghiên cứu.
2) XX cho thấy nhóm huyết thanh chủng phân lập được thuộc vào việc nghiên cứu sự có mặt của các gen tương ứng hoặc việc xác định.
3) Xem Phụ lục F.
4) miliQ là tên thương mại của sản phẩm được cung cấp bởi Millipore Corporation. Thông tin này đưa ra để tạo thuận tiện cho người sử dụng tiêu chuẩn và không ấn định sử dụng sản phẩm này. Các sản phẩm tương tự có thể được sử dụng nếu cho các kết quả tương đương.
5) TaqMan là tên thương mại của sản phẩm được cung cấp bởi Applied Bioystems. Thông tin này đưa ra tạo thuận tiện cho người sử dụng tiêu chuẩn và không ấn định sử dụng sản phẩm này. Các sản phẩm tương tự có thể được sử dụng nếu cho các kết quả tương đương.
6) Vic là tên thương mại của sản phẩm được cung cấp bởi Applied Bioystems. Thông tin này đưa ra tạo thuận tiện cho người sử dụng tiêu chuẩn và không ấn định sử dụng sản phẩm này. Các sản phẩm tương tự có thể được sử dụng nếu cho các kết quả tương đương.
7) Gồm thông tin riêng.
8) Sẽ được xuất bản (soát xét ISO 16140:2003 [17]).


